- Productos
- Catálogos
- News & Trends
- Ferias
Software para secuenciación NGS GensearchNGSde análisisde gestión de datosde diagnóstico
Añadir a mis favoritos
Añadir al comparador
¿Quiere comprar directamente?
Visite nuestra Shop.
Características
- Función
- de análisis, de gestión de datos, de diagnóstico
- Aplicaciones
- para secuenciación NGS, de laboratorio
Descripción
GensearchNGS es una solución de software integrada para el análisis de los datos del DNA-Seq de los equipos NGS de uso común como Roche/454, Illumina, Ion Torrent y Proton. Se ha diseñado y desarrollado en estrecha colaboración con los principales grupos de diagnóstico acreditados en Europa y con el apoyo del proyecto NMDchip FP7.
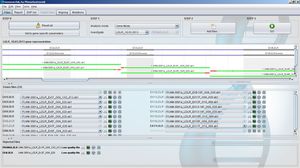
Integra todos los pasos, desde la importación y filtrado de lecturas de secuencias, división de códigos de barras, alineación, detección de variantes y anotación hasta el informe final de las variantes detectadas. Las variantes pueden ser anotadas con varias fuentes de datos públicas y privadas (Ensembl, Alamut, Genome Trax,...) Pueden ser filtradas por combinaciones lógicas entre muestras para estudios familiares.
Características principales:
Un software fácil de usar para resecuenciar los proyectos de NGS en un entorno de diagnóstico, que se ejecuta en sistemas Windows, Linux y MacOS.
Conecta*) a las herramientas de interpretación de variantes, y a bases de datos como: Ensembl, dbSNP, Alamut, Clinvar, Wiki Pathways, Gene Ontology*).
Variantes de filtro entre muestras (por ejemplo, variantes comunes a varias muestras o excluir las variantes de una muestra de otra).
Sistema de base de datos centrada en el paciente para gestionar los datos de la secuencia y las variantes validadas.
Posibilidad de personalizar la exportación de datos al sistema de gestión de su laboratorio.
Posibilidad de publicar sus variantes en Clinvar*).
Desarrollado junto con laboratorios acreditados.
Para proyectos más grandes (WES/WGS): posibilidad de usar varios PCs en paralelo.
Tecnología plugin: alineación con nuestros algoritmos propietarios o públicos como Bowtie, BWA o Stampy.
Anotación y filtrado con descriptores y códigos HPO, OMIM y Orphanet, útiles cuando se buscan mutaciones en exomas o genomas enteros.
---
* Los precios no incluyen impuestos, gastos de entrega ni derechos de exportación. Tampoco incluyen gastos de instalación o de puesta en marcha. Los precios se dan a título indicativo y pueden cambiar en función del país, del coste de las materias primas y de los tipos de cambio.