- Laboratorio
- Medicina del laboratorio
- Software de análisis
- SoftGenetics, LLC.
Software para secuenciación NGS NextGENe®de análisisde laboratorio
Añadir a mis favoritos
Añadir al comparador
Características
- Función
- de análisis
- Aplicaciones
- para secuenciación NGS, de laboratorio
Descripción
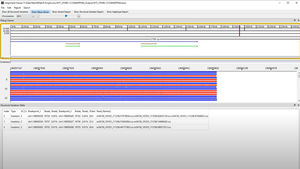
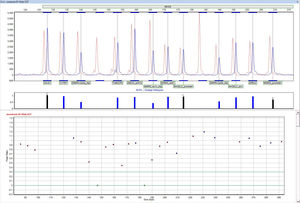
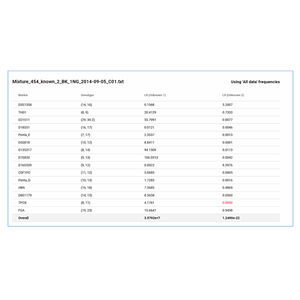
El software NextGENe es el socio analítico perfecto para el análisis de datos de secuenciación de escritorio producidos por los sistemas Illumina® iSeq, Miniseq, MiSeq, NextSeq, HiSeq y NovaSeq, los sistemas Ion Torrent Ion GeneStudio S5, PGM y Proton, así como otras plataformas.
El software NextGENe se ejecuta en un sistema operativo Windows®, que proporciona una interfaz "apuntar y hacer clic" fácil de usar para los biólogos. No requiere secuencias de comandos ni otros soportes bioinformáticos, que suelen ser necesarios cuando se utilizan programas como CLC Genomics Workbench, SeqMan Pro de Lasergene, así como software académico como MAQ & SOAP, Top Hat, BWA & Bowtie.
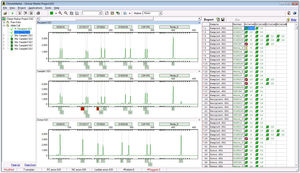
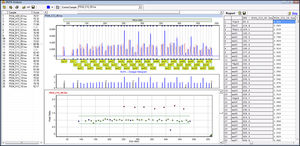
El software NextGENe emplea tecnologías únicas específicas de la plataforma en un paquete multiaplicación independiente. El software NextGENe contiene módulos de análisis para:
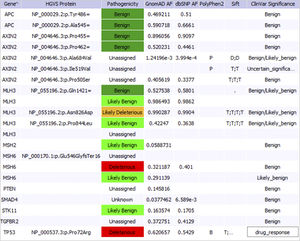
Detección de SNP/Indel
Línea germinal
Somática
Captura del exoma, secuenciación del exoma completo (WES)
Secuenciación del genoma completo (WGS)
Análisis de variantes estructurales (incluida la detección de genes de fusión)
Comparaciones entre familias y tríos
Comparaciones tumor-normal
Detección de variaciones en el número de copias (CNV) y alternativas al cribado tipo MLPA
ensamblaje de novo
Transcriptoma; análisis de splicing alternativo y niveles de expresión de transcritos
ChIP-Seq, expresión génica digital (DGE), análisis y cuantificación de miARN y metagenómica
El software NextGENe está diseñado en un entorno Windows® fácil de usar para los biólogos, lo que reduce significativamente la necesidad de recursos y costes bioinformáticos adicionales. El software NextGENe utiliza hardware de bajo coste basado en el sistema operativo Windows de 64 bits. (Haga clic aquí para ver la configuración de hardware sugerida)
---
Búsquedas asociadas
- Software de análisis
- Software visualizador
- Software de control
- Software de laboratorio
- Software de monitorización
- Software automatizado
- Software de diseño
- Software con sistema de seguimiento
- Software de importación
- Software servidor
- Software para la investigación
- Software de interpretación
- Software de biología molecular
- Software de genética
- Software para secuenciación NGS
- Software NGS
- Software para la investigación clínica
- Software para la genómica
- Software para secuenciación
- Software para secuenciación de ADN
* Los precios no incluyen impuestos, gastos de entrega ni derechos de exportación. Tampoco incluyen gastos de instalación o de puesta en marcha. Los precios se dan a título indicativo y pueden cambiar en función del país, del coste de las materias primas y de los tipos de cambio.