- Laboratorio
- Biología molecular
- Software para informes
- JSI medical systems
Software para informes virSEAKde importaciónNGSde biología molecular
Añadir a mis favoritos
Añadir al comparador
¿Quiere comprar directamente?
Visite nuestra Shop.
Características
- Función
- para informes, de importación, NGS
- Aplicaciones
- de biología molecular
- Tipo
- automatizado
Descripción
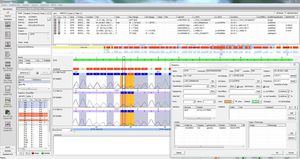
virSEAK (RUO - research use only) ofrece una comparación rápida y cómoda de su secuencia de SARS-CoV-2 con las secuencias disponibles. Después de una configuración personalizada con su kit, simplemente cargue datos fastq o fasta y obtenga una secuencia alineada con variantes.
Además, su secuencia será asignada a un linaje de pangolines (por ejemplo, B.1.1.7) y a un clado GISAID (por ejemplo, L).
virSEAK también comprueba automáticamente si su secuencia contiene las variantes de la proteína pico N501Y y/o E484K.
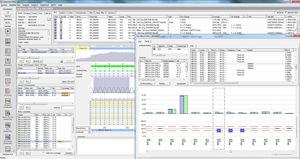
Se muestran los valores de cobertura y calidad, incluyendo: cobertura requerida, absoluta, media y mediana, % de bases "wild type" y "N". Un indicador de calidad muestra si la calidad satisface los umbrales de calidad definidos.
Además, la herramienta está optimizada para la secuenciación NGS de alto rendimiento con funciones automatizadas de importación y exportación y transferencia personalizable de secuencias. Cree fácilmente archivos multifasta para mejorar la presentación de informes a una oficina central de informes para la vigilancia de la situación epidemiológica y la propagación de mutaciones (por ejemplo, para Alemania, el Instituto Robert Koch).
virSEAK - versión instalable:
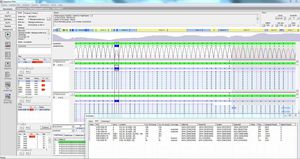
importación sencilla de archivos de secuenciación fastq sin procesar o fasta preprocesados
secuenciación NGS de alto rendimiento con importación y exportación automatizadas por lotes
alerta automática si la(s) mutación(es) N501Y y/o E484K están contenidas
asignación a un linaje de pangolines y a un clado GISAID
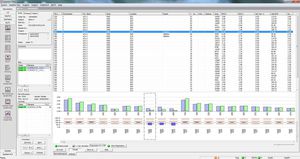
buscar otras variantes con frecuencias
comparar subtipos, recuentos y localizaciones de hallazgos, ...
exportar secuencia y variantes (con meta información, en formato fasta y/o csv)
los resultados se almacenan
secuenciación de alto rendimiento totalmente automatizada con transferencia personalizable de la secuencia
---
Catálogos
No hay ningún catálogo disponible para este producto.
Ver todos los catálogos de JSI medical systemsBúsquedas asociadas
- Software de análisis
- Software para informes
- Software automatizado
- Módulo de software
- Software de importación
- Software servidor
- Módulo de software de análisis
- Software de biología molecular
- Módulo de software de laboratorio
- Módulo de software de captura
- Módulo de software de control
- Software NGS
- Módulo de software de cartografía
* Los precios no incluyen impuestos, gastos de entrega ni derechos de exportación. Tampoco incluyen gastos de instalación o de puesta en marcha. Los precios se dan a título indicativo y pueden cambiar en función del país, del coste de las materias primas y de los tipos de cambio.