- Laboratorio
- Biología molecular
- Módulo de software de análisis
- JSI medical systems
Módulo de software para secuenciación NGS SEQNEXTde análisisde capturade cartografía
Añadir a mis favoritos
Añadir al comparador
¿Quiere comprar directamente?
Visite nuestra Shop.
Características
- Función
- de análisis, de captura, de cartografía
- Aplicaciones
- de laboratorio, para secuenciación NGS
Descripción
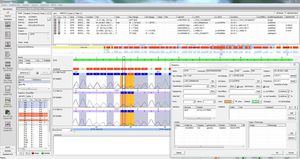
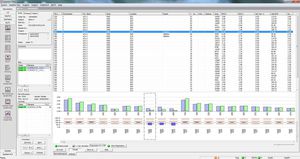
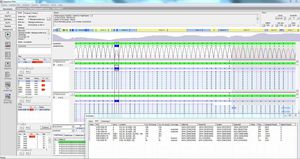
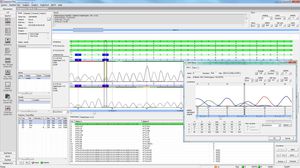
SEQNEXT es una aplicación potente y fácil de usar para el mapeo, alineamiento y detección de variantes de datos de Next Generation Sequencing. SEQNEXT puede analizar datos de todas las plataformas y kits de secuenciación comunes (basados en PCR, enriquecimiento, captura de secuencias). La visualización de todas las variantes detectadas como deleciones, inserciones e indels de cualquier longitud, SNPs, regiones repetidas así como CNVs y fusiones génicas es clara e intuitiva.
SEQNEXT ofrece acceso a bases de datos SNP públicas como dbSNP, 1000 Genomes, ClinVar, ExAC y gnomAD para clasificación y filtrado. Todos los datos de los resultados pueden intercambiarse con nuestra base de datos de variantes para la Experiencia y el Conocimiento Compartidos - varSEAK, transferirse a los Sistemas LIM internos del laboratorio y/o emitirse como informes personalizados de pacientes.
NUEVO Análisis mejorado de datos de secuenciación de virus, por ejemplo, SARS-CoV-2
Trioanálisis y Poolanálisis mejorado
Anotaciones de Ontología de Secuencias en la tabla de variantes
Opción de salto a varSEAK y a todas las BD externas disponibles como dbSNP, ClinVar, UK10K, ESP, gnomAD y ExAC
Análisis de secuenciación del exoma completo (WES)
Compatible con datos de todas las plataformas y kits habituales de secuenciación de próxima generación
Fácil configuración de regiones objetivo individuales (ROI) mediante la importación de archivos de cama o de manifiesto basados en hg19 y/o hg38
Importación de listas de genes para filtrado y análisis de datos WES
Análisis de archivos fastq, fastq.gz o bam
Estándares eficientes para mapeo, alineación, calidad y llamada de variantes (algoritmos utilizados: BWA y Smith-Waterman, adaptados por JSI)
Ajustes personalizados para el análisis y la llamada de variantes (≥ 0,1 %)
Alta sensibilidad y especificidad para la detección de deleciones, inserciones e indels de cualquier longitud, SNPs, regiones repetidas, así como CNVs y fusiones génicas
---
Catálogos
SEQUENCE PILOT
2 Páginas
Búsquedas asociadas
- Software de análisis
- Software para informes
- Software automatizado
- Módulo de software
- Software de importación
- Software servidor
- Módulo de software de análisis
- Software de biología molecular
- Módulo de software de laboratorio
- Módulo de software de captura
- Módulo de software de control
- Software NGS
- Módulo de software de cartografía
* Los precios no incluyen impuestos, gastos de entrega ni derechos de exportación. Tampoco incluyen gastos de instalación o de puesta en marcha. Los precios se dan a título indicativo y pueden cambiar en función del país, del coste de las materias primas y de los tipos de cambio.